The work program consists of six projects grouped into three themes that pursue the Lifespan AI vision from different perspectives: Data and Methods (D), Models and Interpretation (M), and Inference and Causality (C). D1 will advance DL strategies to explore and process long-term temporal change based on integration of high-dimensional data from multiple sources; D2 will combine neural networks and mixed-effects models to predict individual health trajectories over the life course; M1 will develop „normalizing flow“ methods to derive joint distributions and conditional densities for health data; M2 will create a cognitive digital twin from everyday human activities to predict change across age groups; C1 will develop time-adaptive, explainable AI methods for recurrent neural networks and event times; and C2 will derive a framework for „causal discovery“ in longitudinal studies, combining different data sets and accounting for nonlinearities.
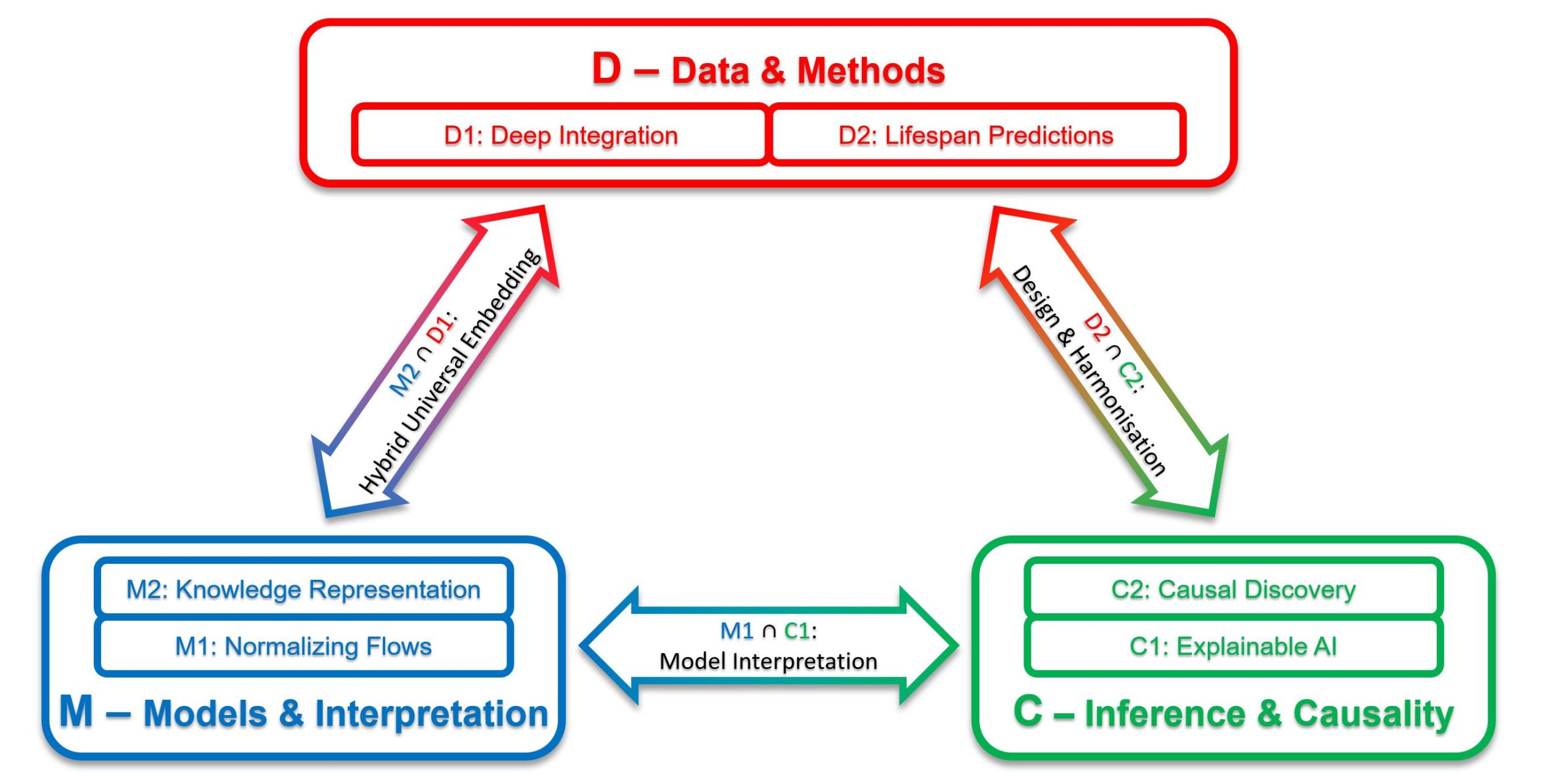
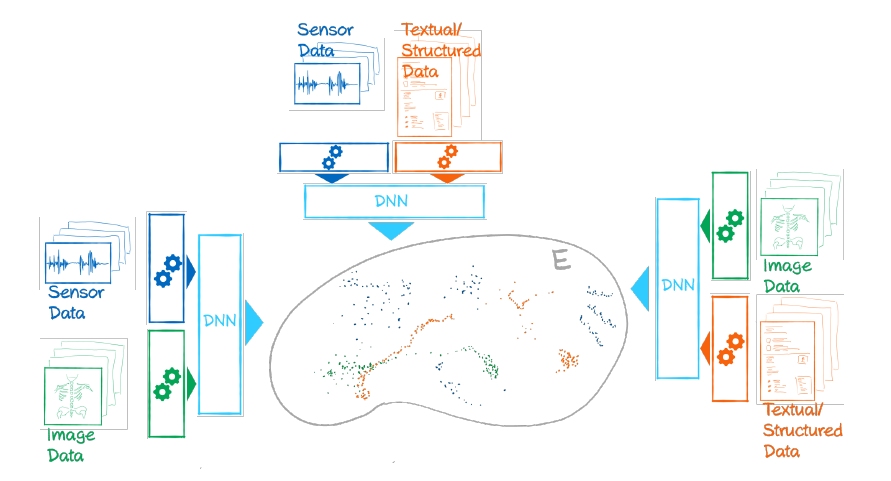
Langzeit-Gesundheitsdaten umfassen das gesamte Leben von Individuen oder Kohorten. Sie stammen aus verschiedenen und veränderlichen Quellen und sind typischerweise lückenhaft und variabel. Die verschiedenartigen Quellen und ihre wechselnde zeitliche Auflösung schon innerhalb einer Kohortenstudie erschweren die computerisierte Integration deutlich. Dies verschärft sich, sollen mehrere Studien gemeinsam ausgewertet werden, die nur einige der Quellen gemein haben und zeitlich nur teilweise überlappen. Um solche Daten für computerbasierte Auswertung zu integrieren, wird das vorliegende Projekt die beschriebenen Hindernisse adressieren. Die entwickelten Methoden auf Basis von Deep-Learning-Ansätzen sollen dabei ein hohes Maß an Generalisierbarkeit und Anwendbarkeit erreichen. Wir werden lernbare und einfach übertragbare Embedding-Modelle einsetzen, die auf zeitaufgelösten sogenannten „Einheiten“ basieren, die eine oder mehrere Datenquellen umfassen. Konzeptuell baut dieser Ansatz auf sogenannten Attention-Modulen auf, die aus Transformer- Architekturen zum Beispiel in der Sprachverarbeitung und Übersetzung bekannt sind. Die entstehenden Embeddings müssen robust generalisieren und sich für „transfer-learning“-Ansätze eignen. Dass dies erreicht wird, stellen wir durch die Verwendung einzigartiger Datensätze sicher, die dem Projekt zur Verfügung stehen werden. Die im Projekt genutzten Datensätze aus zwei großen deutschen Bevölkerungsstudien umfassen eine große Bandbreite an zeitlichen Auflösungen und Datentypen, etwa Fragebögen, Sprachaufnahmen und medizinische Bilder. Unsere Bibliothek trainierter Embedding-Modelle für diese Quellen soll leicht auf neue Daten aus weiteren Studien übertragbar sein. Weiterhin werden wir anhand spezifischer Anwendungsfälle demonstrieren, wie sich Embedding-Räume eignen, um verschiedene Vorhersagen zu unterstützen. Das können zum Beispiel ergänzte Daten zu einem Zeitpunkt sein, fehlende Zeitpunkte einer oder mehrerer Datenquellen, oder sogar Vorhersagen zukünftiger Entwicklungen. Verlässliche Vorhersagen werden von bestimmten Eigenschaften der Embedding-Räume abhängen. Daher ist es ein wichtiges wissenschaftliches Ziel des Projektes, bedeutungsvolle Metriken zu erforschen, um Embedding-Räume quantitativ zu charakterisieren, zum Beispiel hinsichtlich der Datenqualität, Ausreißer, Bias, und anderer Inkonsistenzen, die die prädiktive Kapazität limitieren können. Die Ergebnisse des Projekts haben das Potential, die Datenwissenschaften im Bereich der Gesundheitsdaten maßgeblich zu beeinflussen, denn hier sind zeitaufgelöste Daten mit einer großen Variabilität hinsichtlich der Datentypen und -Qualitäten die Regel. Da unser Ansatz grundlegend unterschiedliche Datentypen und Informationsquellen mit Veränderungen über lange Beobachtungszeiträume vereinigt, erwarten wir, dass die Ergebnisse auch auf Bereiche jenseits der Gesundheitswissenschaften übertragbar sein werden.
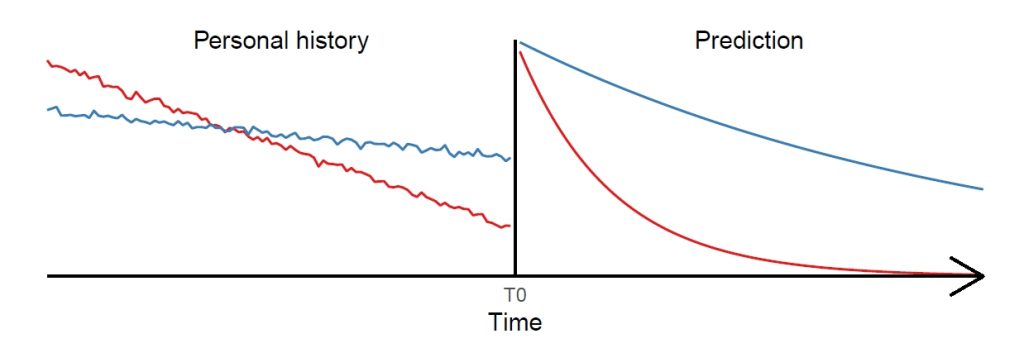
Chronische Krankheiten wie kardiovaskuläre Erkrankungen oder Demenz entstehen typischerweise über lange Zeitspannen im Lebenslauf. Die Untersuchung von frühen Einflüssen auf solche Erkrankungen sowie die Vorhersage von Krankheiten lange vor Krankheitsausbruch stellen die epidemiologische Forschung vor große Herausforderungen. Da es nahezu keine Studie gibt, die die gesamte Lebensspanne abdeckt, bietet die gemeinsame Auswertung mehrerer Kohorten, die verschiedene Lebensphasen abdecken, den vielversprechendsten Ansatz, um gesundheitliche Spätfolgen in realistischen Zeiträumen erforschen zu können. Aktuell gibt es jedoch keine statistische Methode, die es ermöglicht, das komplexe Zusammenspiel verschiedener Faktoren basierend auf den gepoolten Daten mehrerer Kohorten adäquat zu modellieren und vorherzusagen. Diese Forschungslücke soll im Rahmen dieses Projektes geschlossen werden, indem „Lifespan AI“ Methoden entwickelt werden, die individuelle Krankheitsverläufe über lange Zeiträume vorhersagen können. Generalisierte gemischte Modelle (GLMM) bieten ein flexibles, statistisches Tool zur Modellierung von longitudinalen und geclusterten Daten. Die restriktiven parametrischen Annahmen stellen allerdings einen großen Nachteil dar. Flexible nicht-lineare Methoden des maschinellen Lernens wie Random Forests (RF) und tiefe neuronale Netze (DNN) weisen diese Nachteile nicht auf, nehmen aber die Daten implizit als unabhängig und identisch verteilt an. Dies führt in einem Längsschnitt-Setting zu ineffizienten Schätzungen. Um die Stärken der jeweiligen Methoden zu bündeln und Schwächen zu beheben, werden in diesem Projekt sogenannte Mixed-Effects-Machine-Learning (ME-ML) Ansätze vorangetrieben. Dabei wird die Struktur der zufälligen Effekte der GLMM in RF sowie NN integriert. Wir werden ME-ML Ansätze erweitern, um individuelle gesundheitliche Verläufe basierend auf den gepoolten Daten verschiedener Kohorten vorhersagen zu können und die Zeitspanne abschätzen, die basierend auf den entwickelten Methoden valide über den eigentlichen Beobachtungszeitraum hinaus vorhergesagt werden kann. Zudem untersuchen wir Ansätze zur Harmonisierung von Daten verschiedener Kohorten und wie sich die Designeigenschaften von gepoolten Kohortendaten auf die Identifizierbarkeit und Performance von Standardmethoden für Vorhersagen und auf kausale Modellselektion im Lebenslauf auswirken. Zusammengefasst werden theoretische und praktische Untersuchungen in Bezug auf Datenharmonisierung und die Designeigenschaften von gepoolten Kohortenstudien durch die methodische Entwicklung von ME-ML Ansätzen (WP2) komplementiert. Abschließend wird die Vorhersagekraft der neu entwickelten Methoden mit denen von Standardmethoden verglichen. Zudem werden alle Verfahren unter Berücksichtigung verschiedener Kriterien bewertet. Zur Illustration und Validierung der verschiedenen Ansätze werden Daten von verschiedenen Kohortenstudien verwendet.
In den Gesundheitswissenschaften werden statistische Modelle verwendet, um die zeitliche Entwicklung von Gesundheitsendpunkten zu quantifizieren und um zu verstehen, wie diese von Risikofaktoren und/oder Behandlungen beeinflusst werden. Dabei ist die Quantifizierung von Unsicherheiten ein entscheidender Beitrag der Statistik. Trend und Unsicherheit werden am besten durch statistische Verteilungen beschrieben. So liefern Modelle für die gemeinsame Verteilung aller Variablen über alle betrachteten Zeitpunkte – zumindest im Prinzip – alle Schlussfolgerungen, die mit Statistik ohne unüberprüfbare Annahmen möglich sind. Ihre Schätzung ist allerdings eine Herausforderung und benötigt bei hochdimensionalen Daten Methoden der künstlichen Intelligenz (KI). In lebensüberspannenden Gesundheitsdaten sind die Variablen zudem unterschiedlich skaliert, werden oft an individuellen Zeitpunkten gemessen und entstammen verschiedenen, sich nur teilweise überlappenden Datenquellen oder Studienkohorten mit zeitlich-veränderlichen Variablen-Sets. In diesem Projekt sollen diese Schwierigkeiten durch Entwicklung neuartiger sog. Normalizing Flows überwunden werden, die auf invertierbaren residualen neuronalen Netzwerken basieren und verallgemeinerte gemischte lineare Modelle (GLMM) als Basisverteilungen verwenden. Dabei ist von Vorteil, dass GLMMs in den Gesundheitswissenschaften oft angewendet werden. Die Verwendung invertierbarer neuronaler Netze hat den Vorteil, numerisch-analytische Ausdrücke für die gemeinsame Verteilung zu liefern, was die Ableitung statistischer und wissenschaftlicher Aussagen stark vereinfacht. Unser neuer Ansatz basiert darauf, dass zunächst die bedingte Verteilung jedes Merkmals in Abhängigkeit von den anderen Merkmalen durch nichtlineartransformierte GLMMs modelliert wird. Die so gewonnenen bedingten Verteilungen werden dann zu einer Gesamtverteilung durch Mittelung von zufällig-sequenziellen Faktorisierungen der bedingten Verteilungen zusammengefasst. Diese Faktorisierungen können durch Reduktion des Variablen-Sets, auf das bedingt wird, erreicht werden. Zusätzlich werden Algorithmen entwickelt, mit denen ein mit nur vollständigen Beobachtungen geschätztes Gesamtverteilungsmodell mit unvollständigen Beobachtungen angepasst und um neue Merkmale erweitert werden kann. Aus dem Gesamtverteilungsmodell werden schließlich Schätzungen von interessierenden bedingten Verteilungen abgeleitet, aus denen wiederum Punkt- und Intervallvorhersagen abgeleitet werden können. Wir werden zudem Methoden zur Interpretation des mittels KI geschätzten Verteilungsmodells und zur Beurteilung seiner internen und externen Validität entwickeln. Ansätze zur Quantifizierung der Modellanpassung und Berücksichtigung der Modellunsicherheit in den Vorhersageintervallen werden ebenfalls untersucht. Alle Methoden werden auf vom BIPS gepflegte bzw. erhobene Daten der IDEFICS/I.Family-Kohorte, der NAKO-Gesundheitsstudie und von GePaRD angewandt und mit diesen illustriert.
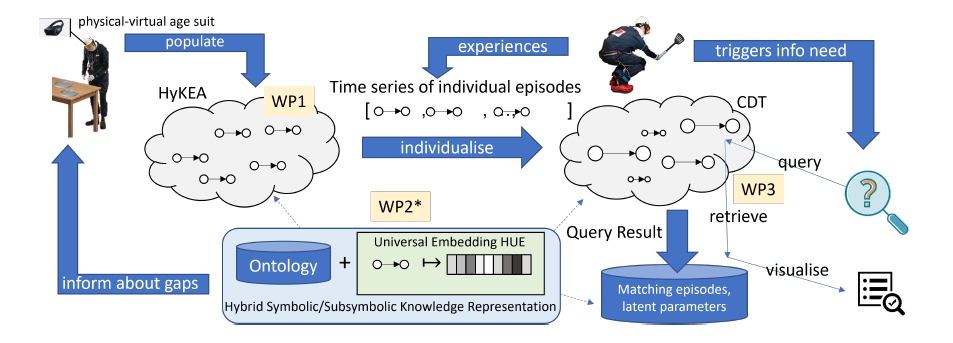
Dieses Projekt untersucht Methoden für die Repräsentation, Inferenz und Modellierung von Fähigkeiten und krankheitsbedingten Einschränkungen einzelner Personen bei der Ausführung von Alltagsaktivitäten. Unser Ziel ist es, Vorhersagen machen zu können, ob bzw. welche Unterstützung eine Person benötigt, wie sich ihre Einschränkungen über die Zeit entwickeln, und wie dies von Kontextfaktoren, wie der Gestaltung der Umgebung oder Müdigkeit, abhängt. Um dies zu erreichen, entwerfen, implementieren und untersuchen wir in diesem Projekt die Prinzipien einer „Lifespan Predictive Modelling Engine“ (L-PME), einer hybriden Wissensrepräsentation und einem Framework zur Inferenz, das Personen begleitet, ihre Alltagsaktivitäten aufzeichnet und individuelle Einschränkungsmodelle in einem lebenslangen Prozess aufbaut. Um eine Person zu repräsentieren, schneidet L-PME einen individuellen „cognitive digital twin“ zu auf der Basis einer generischen, Einschränkungs-bewussten Wissensrepräsentation von Alltagsaktivitäten, die folgendes erfasst, um offene Anfragen zu beantworten und Aufgabenerfolg und Verhalten vorherzusagen: (1) die zeitliche Entwicklung von krankheitsbedingten Einschränkungen im Kontext veränderlicher Situationen, Bedürfnisse und Aufgaben, (2) den kausalen Einfluss von Fähigkeiten und Einschränkungen auf Manipulationsaufgaben im Alltag und (3) die latente Verknüpfung von Verhaltensweisen in verschiedenen Alltagsaktivitäten. Wir werden die Grundlagen der Methoden zur Repräsentation, Inferenz und Modellierung untersuchen, indem wir sie auf Personen mit konfigurierbaren physisch-virtuellen Alterungsanzügen bei der Durchführung von Alltagsaktivitäten, wie etwa Tischdecken, anwenden. Die Verwendung solcher Alterungsanzüge erlaubt es uns, die grundlegenden Prinzipien der L-PME unter beobachtbaren und kontrollierbaren Bedingungen zu untersuchen, indem die Parameter des physisch-virtuellen Alterungsanzugs systematisch variiert werden.
Die jüngsten Erfolge der künstlichen Intelligenz (KI) in fast allen Forschungsfeldern werden flankiert von Methoden der erklärbaren KI (XAI), die versuchen, KI-Systeme und Black-Box-Modelle des maschinellen Lernens zu erklären. Diese Methoden haben sich in vielen Forschungsfeldern bewährt, auch bei der Anwendung auf Gesundheitsdaten. So werden XAI-Methoden beispielsweise zur Erkennung relevanter Pixelregionen in der medizinischen Bildgebung eingesetzt. Bei lebensüberspannenden Gesundheitsdaten haben wir es jedoch mit heterogenen Datenquellen mit unterschiedliche Datenstrukturen und Dimensionen, mit longitudinalen Prädiktoren, die über lange Zeiträume gemessen werden, und zeitabhängigen Ereignissen zu tun. Die Integration solcher Daten in KI-Systeme ist eine besondere Herausforderung. Ebenso herausfordernd ist es jedoch, die resultierenden Modelle zu erklären. In diesem Projekt gehen wir diese Herausforderung an, indem wir XAI-Methoden entwickeln, die Daten über die gesamte Lebensspanne verarbeiten können, einschließlich longitudinaler Prädiktoren, zeitabhängiger Ereignisse und Kombinationen verschiedener Datenquellen, die sich über die Lebenszeit verändern. Wir befassen uns sowohl mit der Interpretation, d.h. dem Verständnis eines Modells an sich, als auch mit der Erklärung, d.h. der Anwendung von Post-hoc-Methoden auf bereits trainierte Modelle. Zur Interpretation verwenden wir invertierbare neuronale Netze, die die Ausgabe des Netzes direkt mit seiner Eingabe in Beziehung setzen. Zur Erklärung entwickeln wir Methoden, die auf sog. „attributions“ (z.B. gradientenbasiert), „attention mechanisms“ und „loss“-basierten Methoden aufbauen. Darüber hinaus entwickeln wir Methoden zur Erzeugung sog. „Knockoffs“ für lebensüberspannenden Gesundheitsdaten mit „Normalizing Flows“, mit dem Ziel hochdimensionale bedingte Variablenselektion und bedingte Unabhängigkeitstests auf solchen komplexen Daten zu ermöglichen. Zusammengefasst zielen wir darauf ab, die in der Forschungsgruppe entwickelten KI-Modelle zu erklären und zu interpretieren. Dies ist von entscheidender Bedeutung für die Forschungsgruppe und ganz allgemein für die Gesundheitswissenschaften, da es zu dem übergeordneten Ziel beiträgt, Gesundheits- und Krankheitsmechanismen zu verstehen. Darüber hinaus spielt unsere Forschung auch eine große Rolle für das Gebiet der KI allgemein, da unsere methodischen Fortschritte die KI über Gesundheitsdaten hinaus bereichern, z.B. bei der Text- oder Sprachverarbeitung.
Unser Projekt hat zum Ziel, innovative und robuste Ansätze zu entwickeln, um mit Hilfe neuer Methoden der künstlichen Intelligenz kausale Zusammenhänge im Lebenslauf aufzudecken und zu quantifizieren. Solche Ansätze werden dringend benötigt, da randomisierte Studien, die als Goldstandard der kausalen Inferenz gelten, für langfristige Fragestellungen über den gesamten Lebenslauf, wie z.B. zur Abschätzung der Auswirkungen des Lebensstils in der Kindheit auf die Gesundheit im Erwachsenenalter, so gut wie nicht existieren. “Causal discovery” (kausale Modellselektion) bezeichnet eine Klasse von Methoden, die von einem multivariaten Datensatz ausgehen (Input) und im Idealfall einen gerichteten azyklischen Graphen ausgeben (Output), der die probabilistische kausale Struktur zwischen den Variablen (als Knoten im Graph) darstellt. Solche Methoden werden zwar allmählich auch im biomedizinischen und sozialwissenschaftlichen Bereich angewandt, aber es gibt bisher keine Beispiele dafür, dass mehrere zeitlich strukturierte Datensätze kombiniert wurden, um eine große Spanne im Lebenslauf abzudecken – unser Vorhaben wird diese Lücke schließen. Das Projekt wird zunächst die theoretische Grundlage für unsere Forschung legen, indem ein kausaler Selektionsalgorithmus entwickelt wird, der zeitlich strukturierte Datensätze kombinieren kann, so dass eine große Spanne im Lebenslauf abgedeckt werden kann, und der sowohl nachweislich korrekt und vollständig sowie praktisch nützlich ist, und zwar den sogenannten TIOD Algorithmus – “temporal integration of overlapping datasets”. Des Weiteren werden wir moderne maschinelle Lernverfahren hinzuziehen (z.B. „causal generative neural networks“), damit der TIOD Algorithmus auch für Situationen geeignet ist, die nicht-lineare und nicht-parametrische Modellierungen erfordern. In diesem Zusammenhang muss ein besonderer Fokus auf die Rolle von Studiendesigns für Multi- Kohorten gelegt werden, da manche Designs informativer sein können als andere. Zudem werden wir Methoden zur Quantifizierung kausaler Beziehungen, wie sie von TIOD gefunden werden, auf den Fall verallgemeinern, dass auch langfristige Effekte aus getrennten Datenquellen geschätzt werden können. Um unseren methodischen Ansatz zu validieren und die praktische Nützlichkeit zu demonstrieren, werden wir im Verlauf des Projekts relevante epidemiologische Forschungsfragen eruieren und entsprechende Datensätze aufbereiten, z.B. indem wir die IDEFICS/I.Family Kohortenstudie und die NAKO Gesundheitsstudie kombinieren. Hierbei wird die Datenharmonisierung eine eigene Herausforderung darstellen. Die Anwendung von TIOD auf diese Daten soll letztlich neue Einsichten liefern, z.B. in die kausalen Zusammenhänge zwischen Faktoren in der Kindheit und Gesundheit im Erwachsenenalter.
Die Universität Bremen stellt zwei Brückenpositionen für Lifespan AI zur Verfügung.
Eine Stelle für einen Disziplin-Connector:
Eine Stelle für einen Datenverwalter:

